SARS-CoV-2
mRNA-Impfstoffanalytik
Die 4base lab AG unterhält GMP-Labors zur Qualitätssicherung biopharmazeutischer Arzneimittel und ist Teil der Prozesskette zur Herstellung von COVID-19-mRNA-Impfstoffen.
Insbesondere Analytiken zur Qualitäts- und Identitätsbewertung von mRNA-Impfstoffen werden von uns durchgeführt:
- Rückstandsanalytiken zu pDNA und Bakterien-gDNA und RNA
- Kontrolle der Nukleinsäuresequenz in Bakterien-produzierten DNA-templates
- Kontrolle der Sequenz durch cDNA-Sequenzierung
- Sequenzierung der Open Reading Frames in mRNA
- SRCA-Sequenzierung von mRNA
- Analyse der stabilitätsbestimmenden nicht-translatierten 3prime Tail-Längen von mRNA
- Analyse von Impfstoffen durch RNA-Direktsequenzierung
Produkte zur SARS-CoV-2-Analytik
4BLqSARS-CoV-2-RNA-Standard
RNA-Positivkontrolle für die RT-PCR zum Nachweis von SARS-CoV-2. Das RNA-Molekül enthält alle Bindestellen der Primer- und Sondensequenzen der Charité- und CDC-Protokolle. Auch mit den im Juni 2020 durch die Charité aktualisierten RdRP-Primern einsetzbar. (LINK auf Manual)
Betriebliches SARS-CoV-2-Monitoring
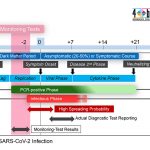
Die Mehrheit der SARS-CoV-2-Infektionen ist auf die Übertragung durch prä- oder asymptomatische Personen zurückzuführen. Die begrenzte Möglichkeit, diesen Träger-Personenkreis vor deren Übergang in eine Phase hoher Virusausscheidung zu erkennen, ist der Hauptgrund für ein Versagen des Covid-19 Managements und des Mitarbeiterschutzes am Arbeitsplatz.
Die 4base lab hat frühzeitig mit dem Aufbau geeigneter Testsysteme im Rahmen eines SARS-CoV-2-Überwachungsplans für Mitarbeiter und Arbeitsumgebungen begonnen, mit dem Ziel, unkontrollierte Ausbrüche während den Arbeiten an den COVID-19 Impfstoffen innerhalb der Belegschaft zu verhindern.
Wir beraten Sie gerne.
SARS-CoV-2-Viren im Abwasser
4base lab weist Corona-Viren im Abwasser nach. Die Untersuchung der kommunalen Abwässer auf Coronaviren kann wertvolle epidemiologische Daten liefern, aus denen Entscheidungen mit gesundheitspolitischer Relevanz getroffen werden können.